Geneious Prime ペアワイズアライメント
Geneious で使用されるペアワイズアライメントは、 Needleman-Wunsch アルゴリズムと Smith-Waterman のアルゴリズムを使いた動的計画法に基づいて計算されます。
ペアワイズアライメントではアルゴリズムの選択に加えて、スコアリングスキームとギャップペナルティの設定にも注意を払う必要があります。スコアリングスキームはアライメントにあるミスマッチや置換に対する感度を制御し、ギャップペナルティはアライメントへのギャップの挿入や伸長に影響を与えます。
このチュートリアルでは、ピグミーチンパンジーの配列とオリジナルの配列を編集したシークエンスを使って、ドットプロット(ドットマトリックス法)とペアワイズアライメントについて学びます。
1. データの取り込み
下記ページよりチュートリアルで使用するデータをダウンロードします。
www.geneious.com
ソースパネルでインポート先のフォルダを選び(ここでは、Local 直下に指定しています)、zipファイルを解凍せずに Geneious Prime にドラッグ&ドロップします。
ソースパネルに Pairwise_alignments フォルダが作成され、ドキュメントテーブルに7つのドキュメントがインポートされていることを確認して下さい。
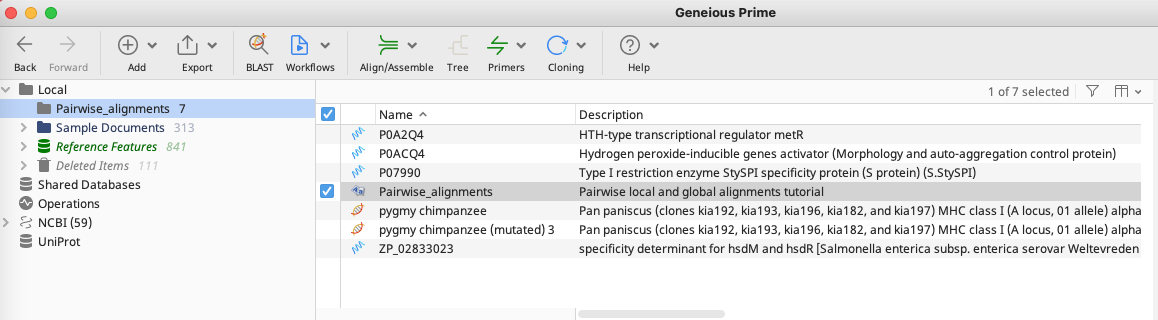
2. Dotplot - ドットプロット
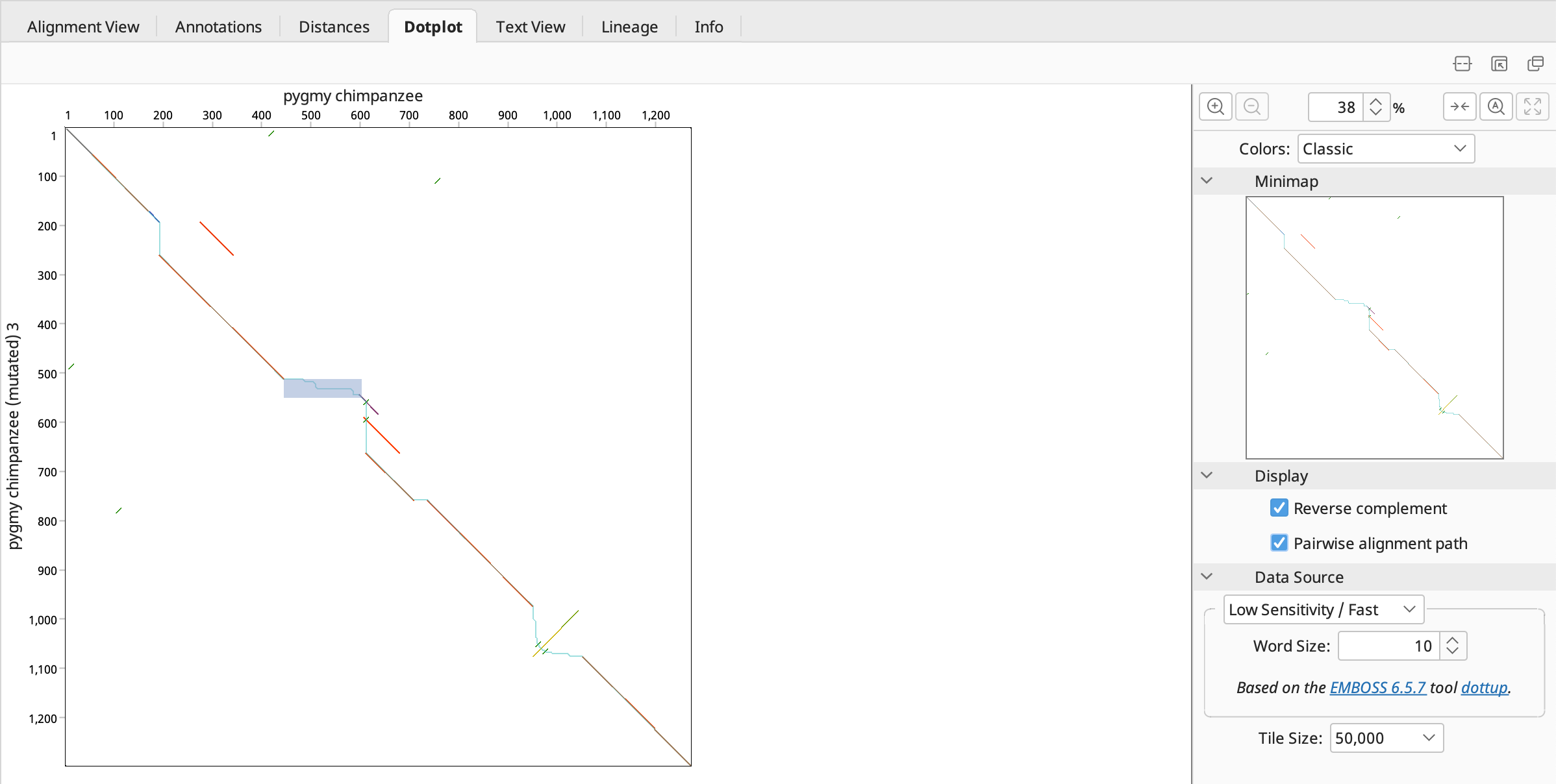
ドットプロット(ドットマトリックス)は、反復配列、反転、転座の確認など行うことができ、配列アライメントだけでは再現できない配列のペア間の類似領域を視覚的に表示します。縦軸(右から左)と 横軸(上から下)に各配列をとり、両軸に全く同じ配列があれば右下がりの対角線が現れます。この対角線に対して平行な線は配列に繰り返しのパターンがあることを示し、左上がりの対角線は相同性のある配列の方向が逆方向になっていることを示します。
ドキュメントテーブルで比較する配列を選択します。この例では![]() pygmy Chimpanzee と
pygmy Chimpanzee と ![]() pygmy chimpanzee (muted) 3 を選択し、ドキュメントビューアの Dotplot タブをクリックします。
pygmy chimpanzee (muted) 3 を選択し、ドキュメントビューアの Dotplot タブをクリックします。
この例では、x軸の上部に pygmy chipanzee (muted) 3 のシークエンス、y軸の左側に pygmy chipanzee のシークエンスが表示された 2Dグリッドが表示れます。全体的な傾向として左上から右下に対角線があるのがわかります。
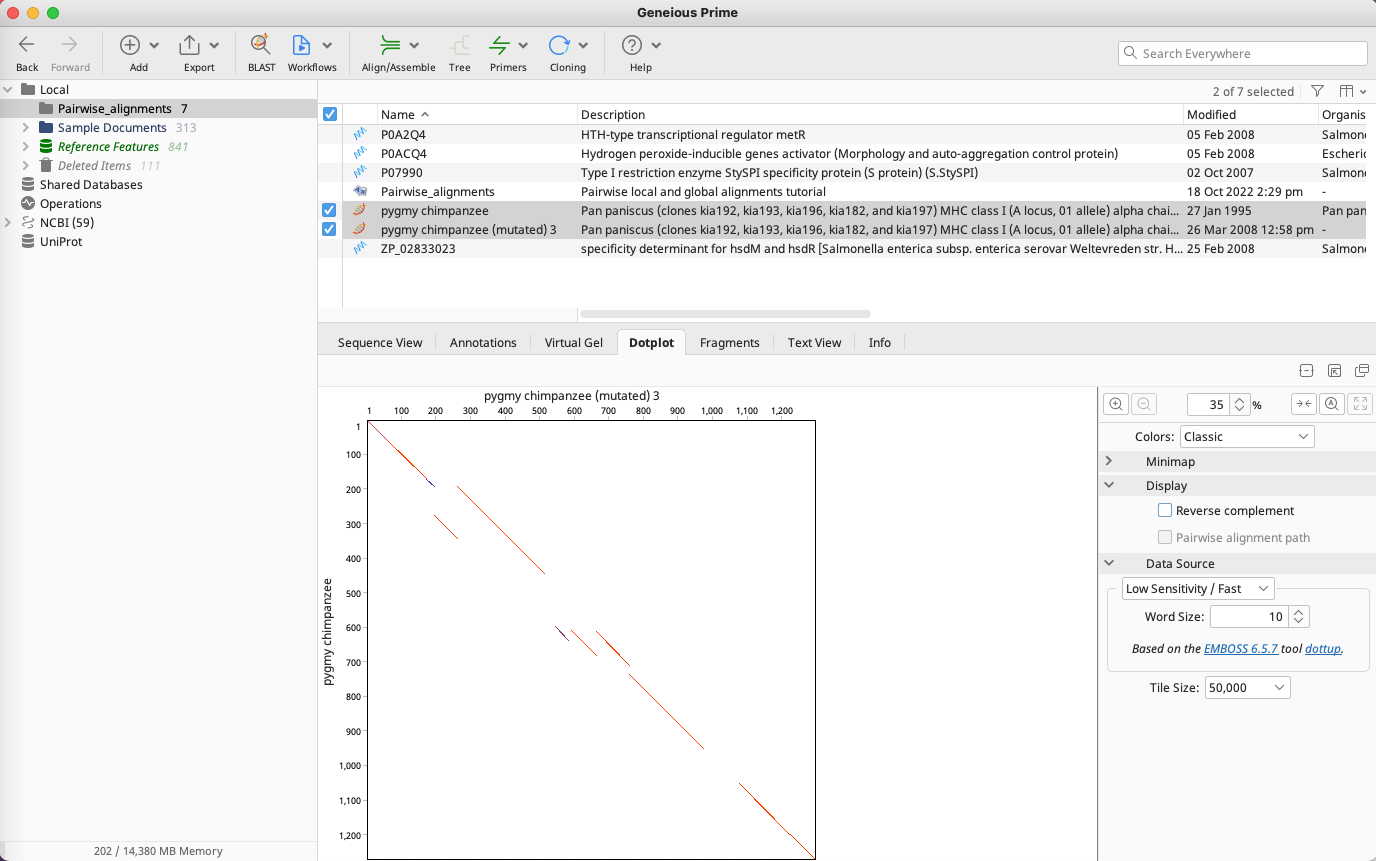
感度調整は、コントロールパネルの Data Source から行います。
ドロップダウンメニューから High Sensitivity / Slow を選択すると、スコアマトリックス、ウィンドウズサイズ、閾値などを設定することが可能です。ウィンドウサイズを大きくすると、小さい対角線間のギャップが埋まり、より長い対角線を作る傾向があります。ウィンドウサイズを小さくすると、対角線は短くなり見える範囲も狭くなります。閾値の設定によって、ドットプロット内のノイズが増加・減少します。
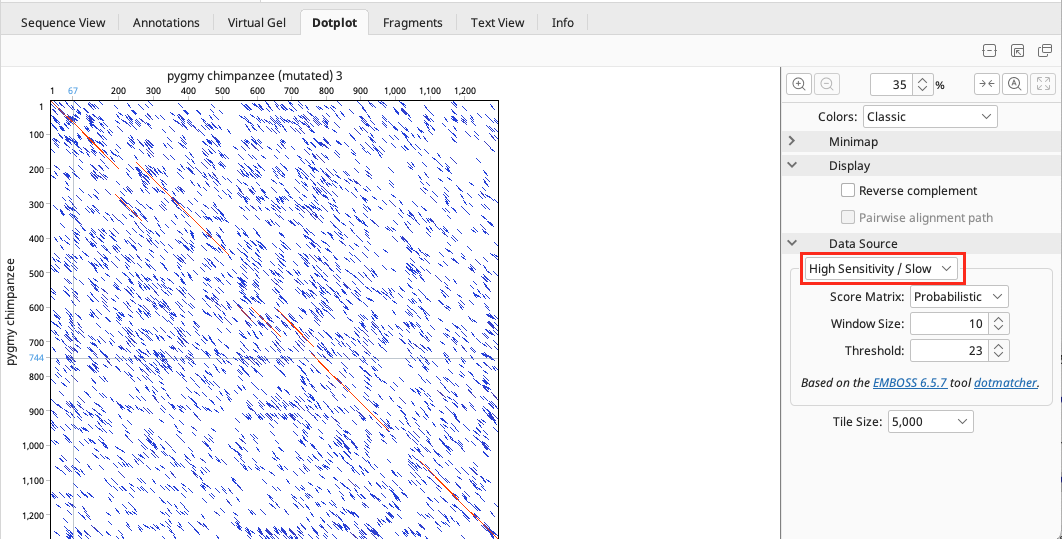
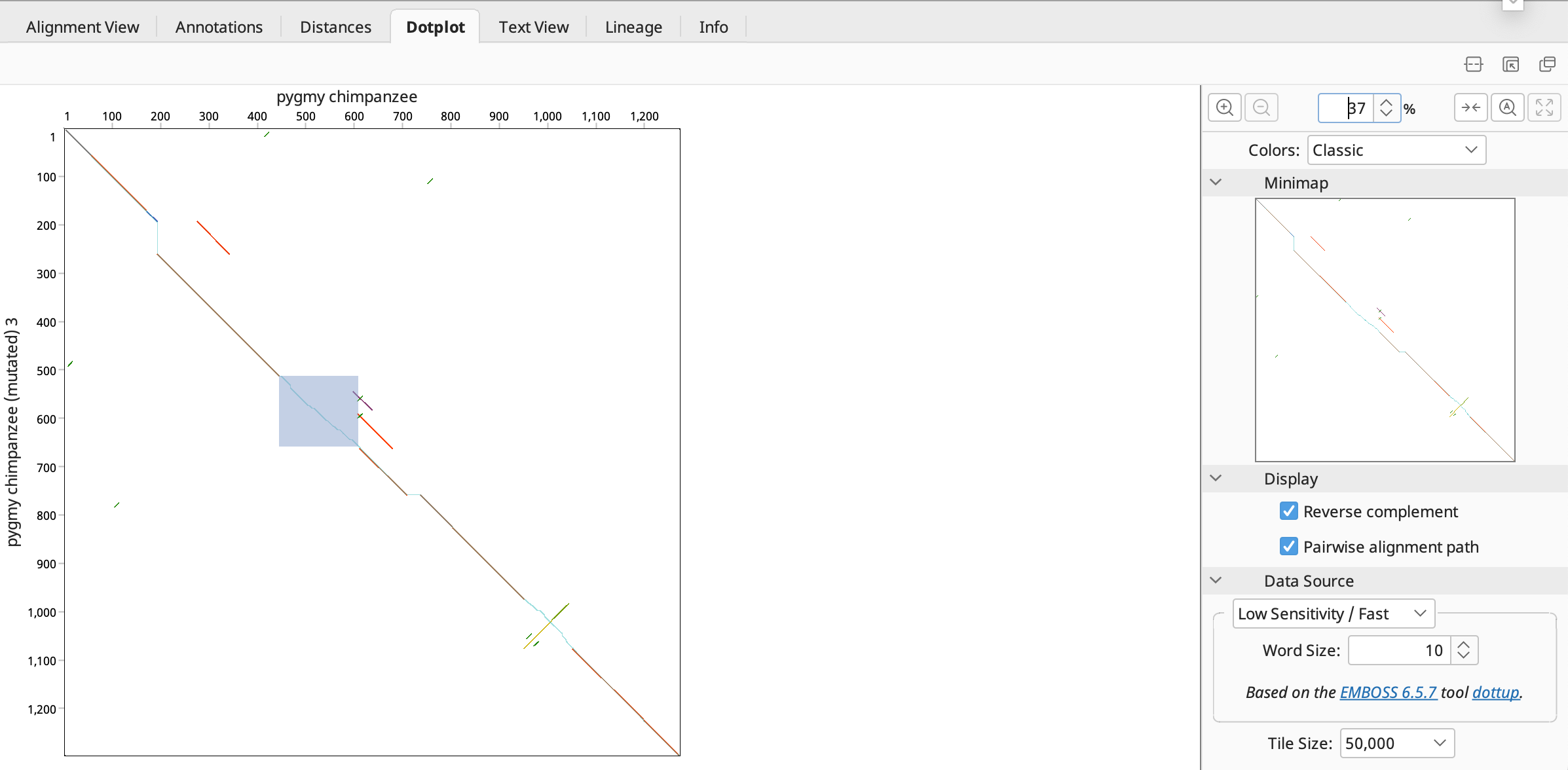
逆位を表示する場合は、Reverse complement にチェックを入れます。
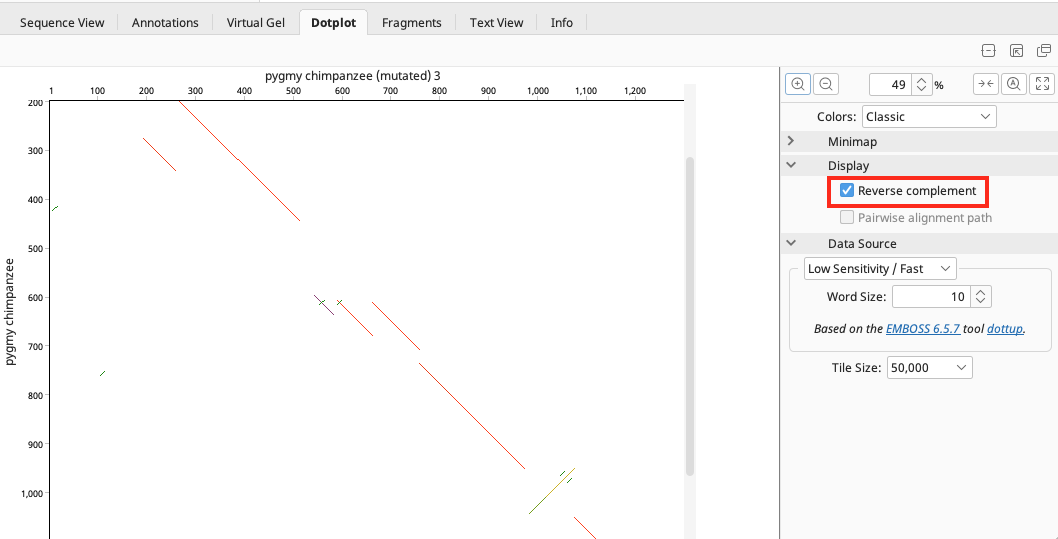
このようにドットプロットは2つの配列がどのように関連しているのか、直感的に傾向をつかむのに有効な方法です。
3. Pairwise Alignment - ペアワイズアライメント
ドキュメントテーブルで ![]() pygmy Chimpanzee と
pygmy Chimpanzee と ![]() pygmy chimpanzee (muted) 3 を選択し、ツールバーの Align/Assemble から Pairewise Align を選択します。
pygmy chimpanzee (muted) 3 を選択し、ツールバーの Align/Assemble から Pairewise Align を選択します。
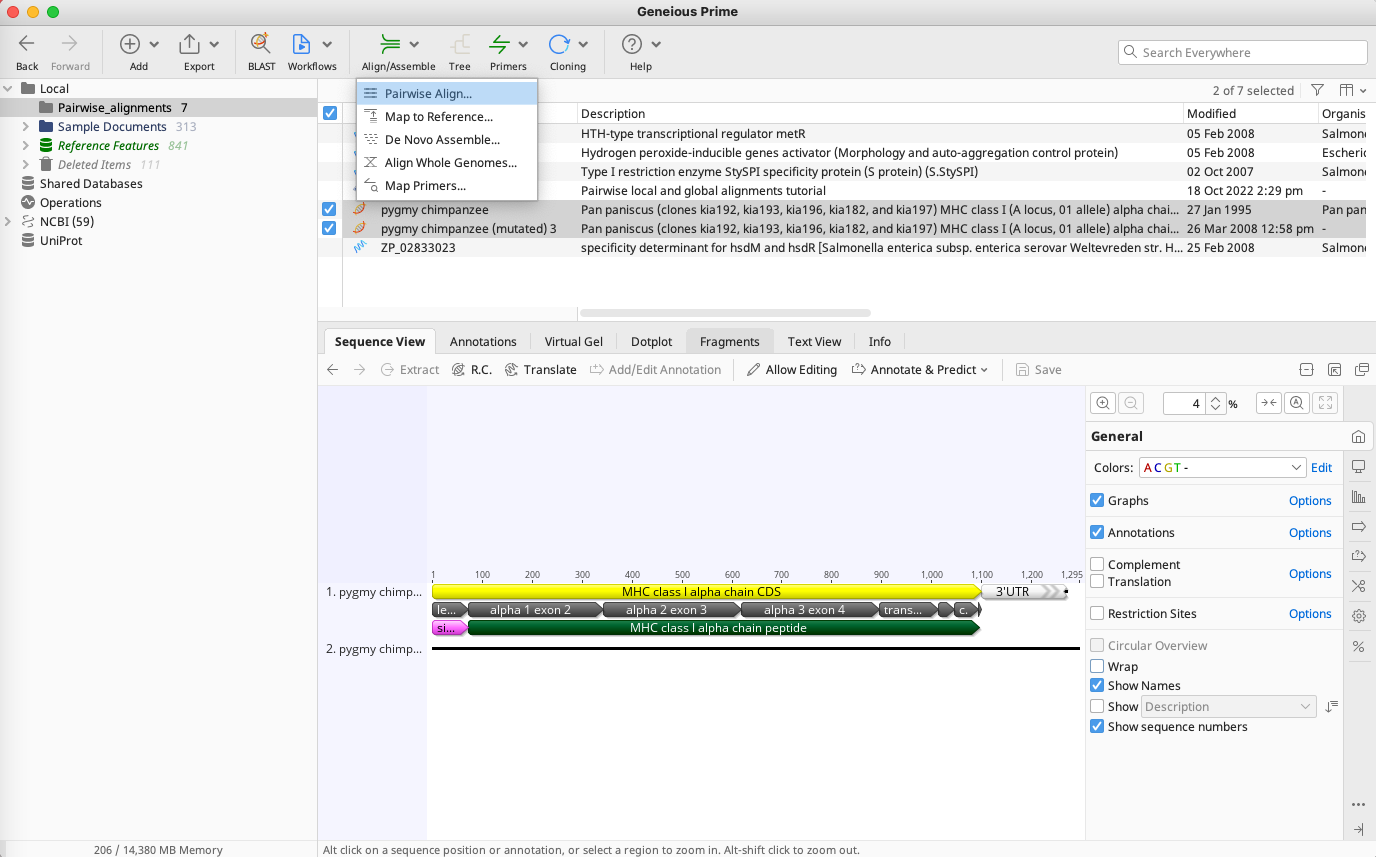
Geneious には、さまざまなアライメント方法が用意されています。このチュートリアルでは Geneious Alignment を使用し、デフォルトのパラメータを使用します。(パラメータをデフォルトに戻すには、 ![]() をクリックし Reset to Defaults を選択します)。
をクリックし Reset to Defaults を選択します)。
Geneious Alignment を選択後、OKボタンをクリックします。
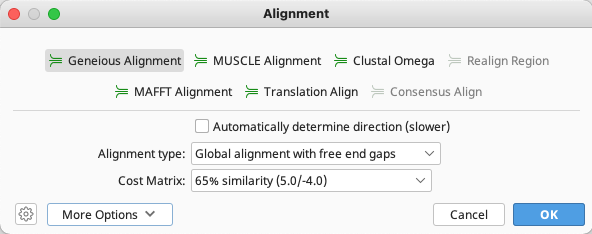
ドキュメントテーブルに ![]() Nucleotide alignment が追加され、またドキュメントビューアの Alignment View にアライメントが作成されます。
Nucleotide alignment が追加され、またドキュメントビューアの Alignment View にアライメントが作成されます。
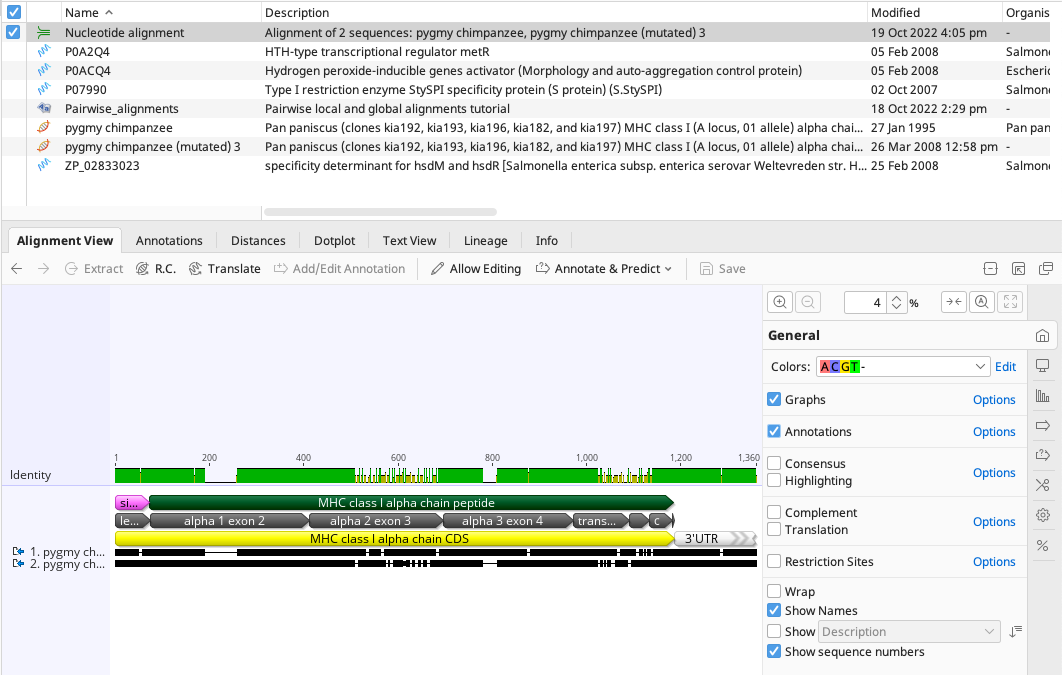
コントロールパネルの Annotations のチェックを外し、Wrap にチェックを入れるとアライメント全体が見やすくなります。Identity 上で不一致のある部分が縞模様になるので、ミスマッチのある領域が簡単に見つかります。
アライメントを見ると、ギャップがほとんどなくミスマッチの多い領域があります。これは、デフォルト設定のギャップペナルティの値が高いことに起因します。
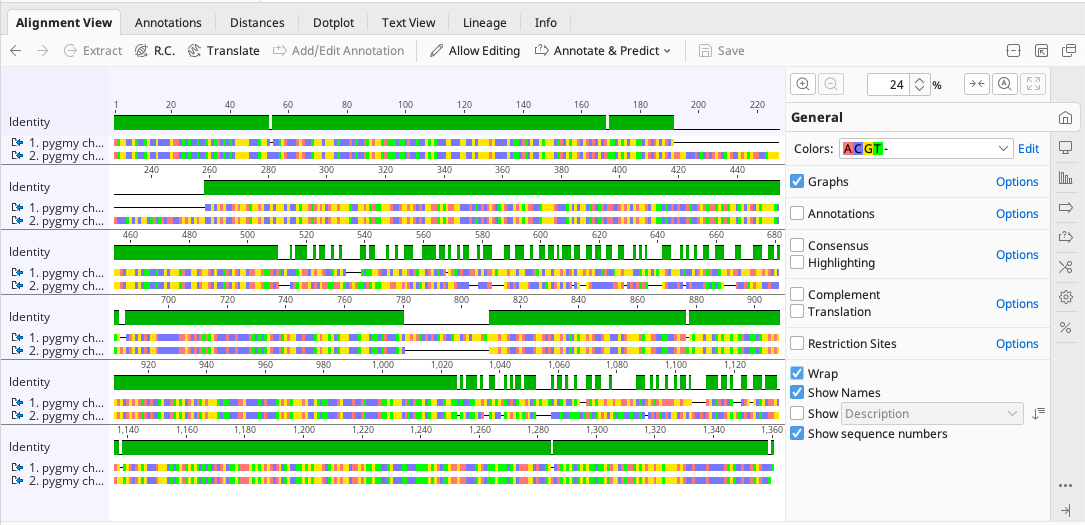
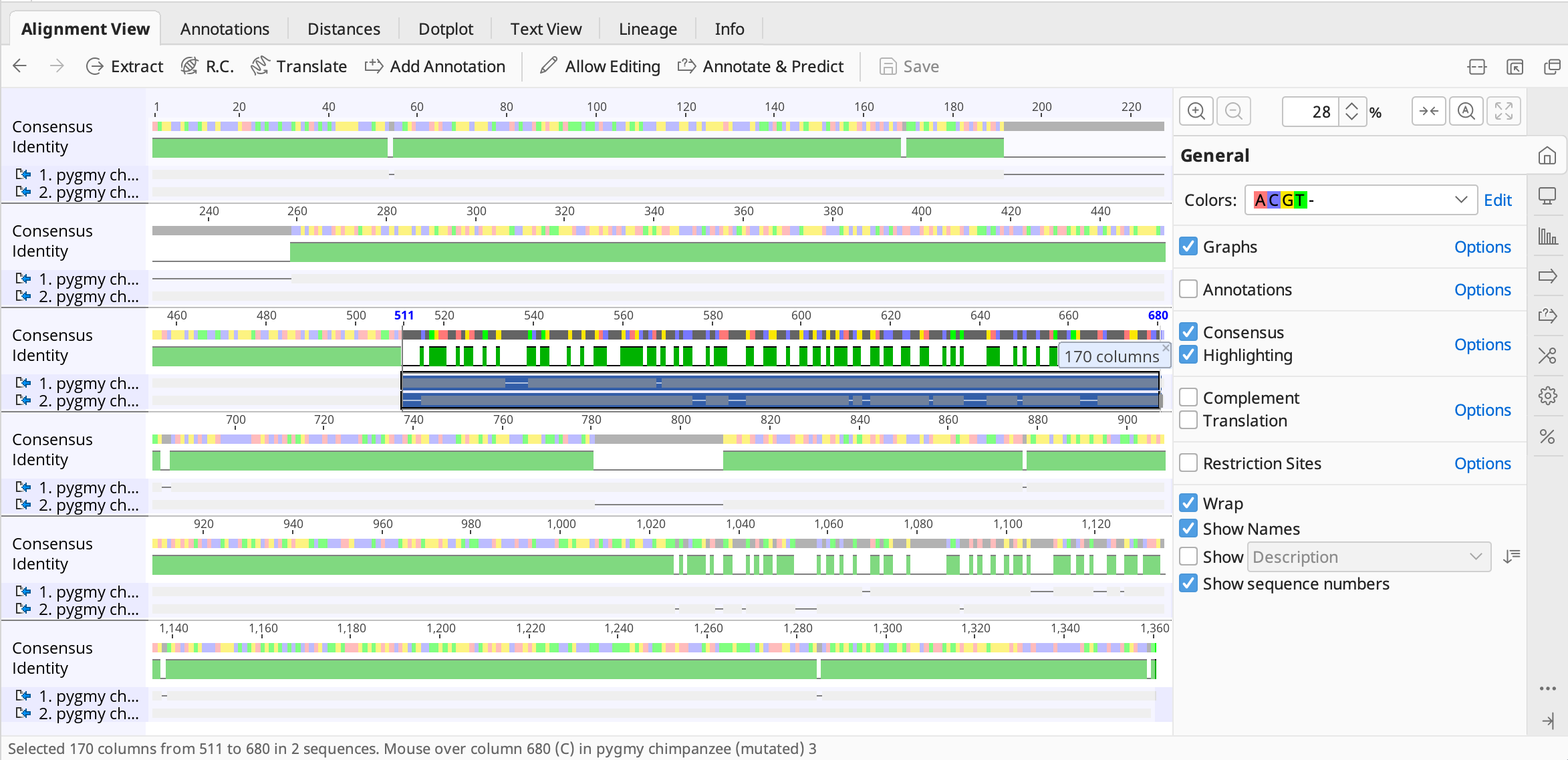
Alignment Viewで、ミスマッチのある最初の領域を見てみましょう。
目的の領域を見つけやすくするため、コントロールパネルの Consensus と Highlighting のチェックを入れ、コンセンサス配列とハイライト表示を有効にします。
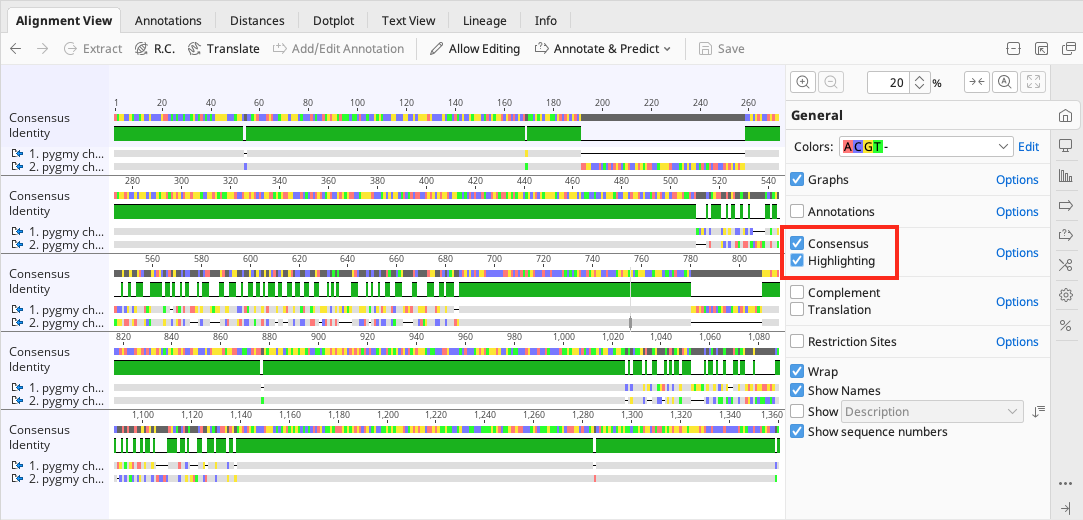
次に、Highlighting のオプションからハイライト表示を Disagreements to Consensus に設定します。
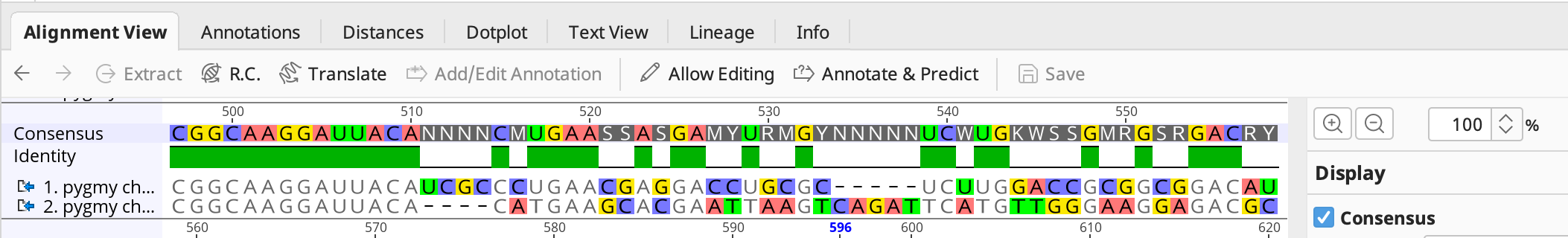
コンセンサス配列と一致するアライメントの塩基はグレーで表示されます。Consensusオプションで Ignore Gaps が選択されていない場合もアライメントの塩基はグレーで表記されます。
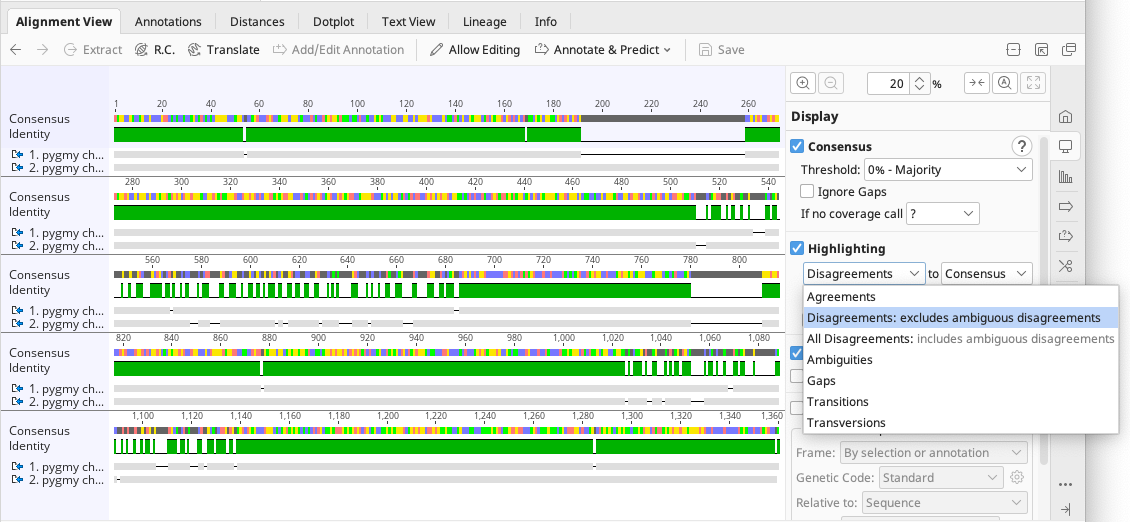
ミスマッチ領域をドラッグ&ドロップで選択すると、選択した領域がドットプロットに反映されます。
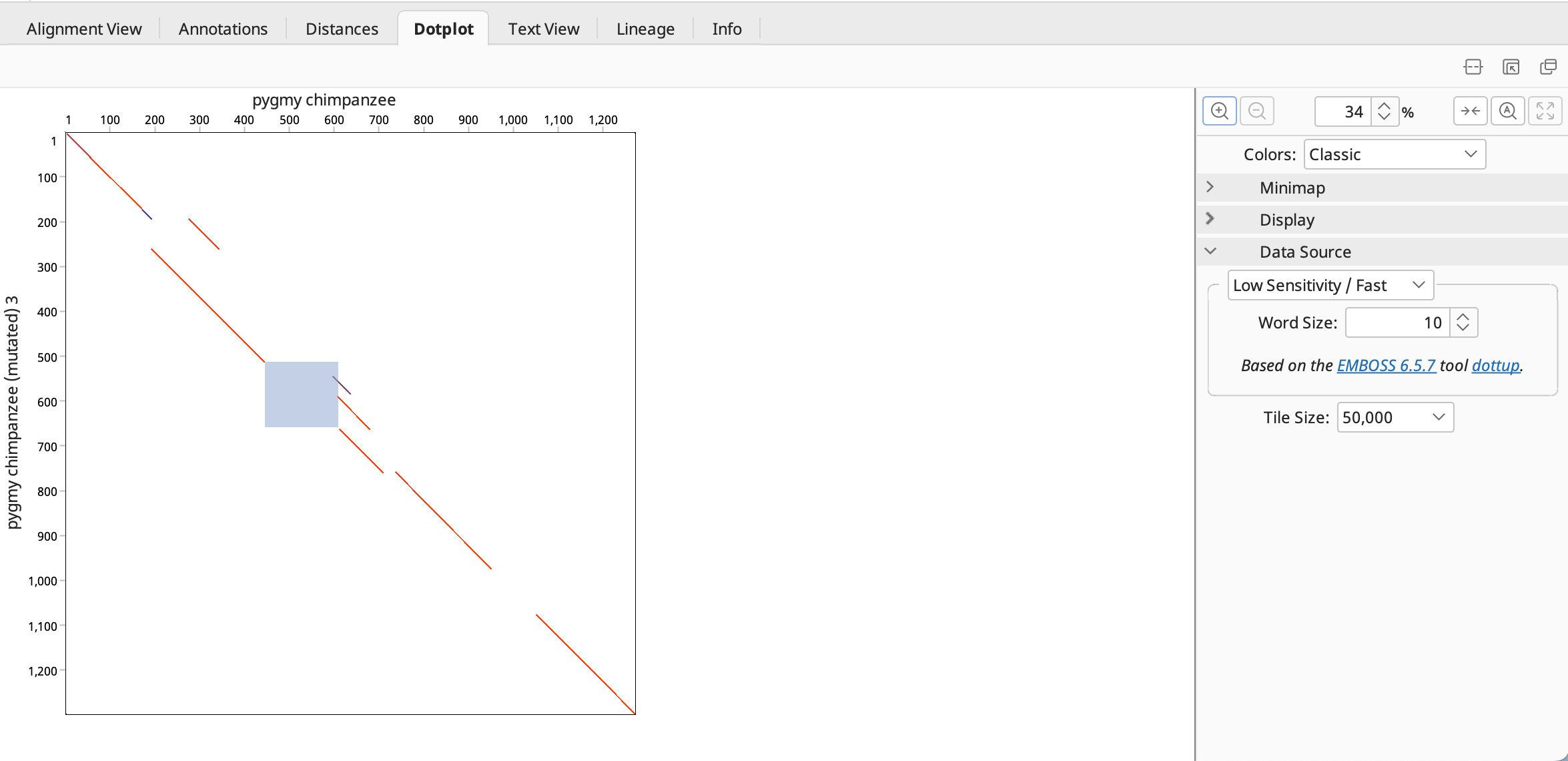
Note: 選択した領域がドットプロットに反映されない場合は、Alignment View で2つの配列が選択されているか確認して下さい。


選択したミスマッチのある領域はオリジナルの配列では 443-609 、変異した配列では 511-660 です。この領域には複数の経路が存在し、数学的に最もコストの低い経路がとられています。しかし、明らかに一致するものがあり真の関係を反映しているとは言えず、設定を修正して再びアラインメントする必要があります。
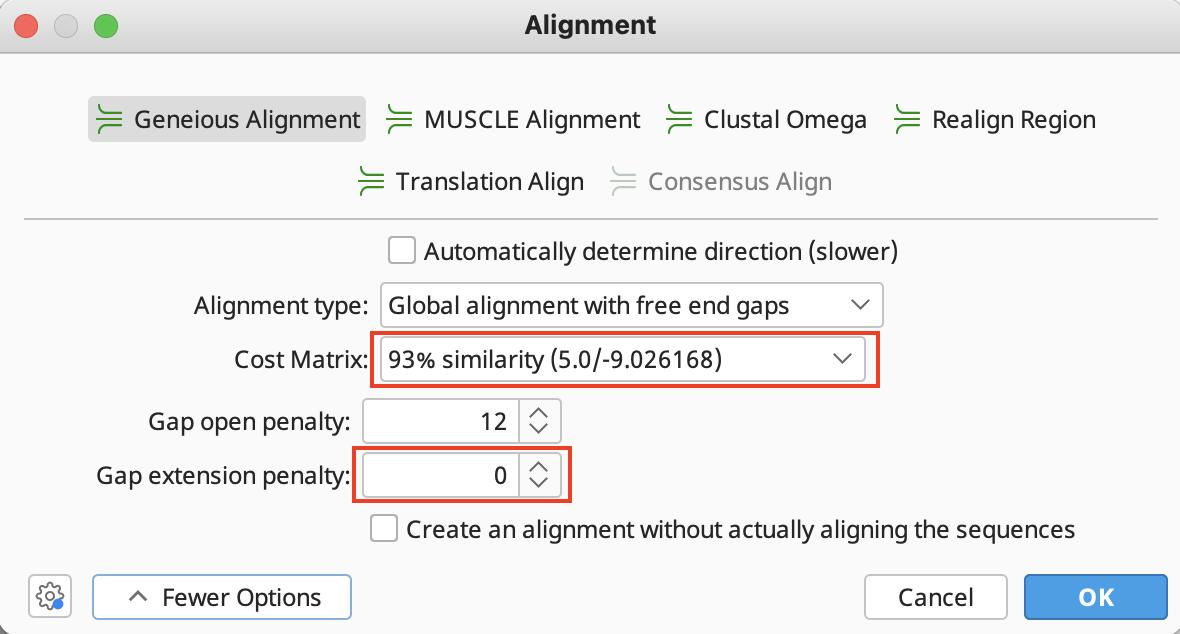
ドキュメントテーブルの ![]() Nucleotide alignment にチェックを入れ、ツールバーから Align/Assemble→Pairwise Alignを選択します。Alignmentダイアログの More Options ボタンをクリックし、Cost Matrix を 93% similarity 、Gap extention penalty を 0 設定しOKボタンをクリックします。
Nucleotide alignment にチェックを入れ、ツールバーから Align/Assemble→Pairwise Alignを選択します。Alignmentダイアログの More Options ボタンをクリックし、Cost Matrix を 93% similarity 、Gap extention penalty を 0 設定しOKボタンをクリックします。
同じコンセンサスとハイライトのオプションセットでアライメントを見ると、2つの配列の間にほとんど不一致がないことがわかります。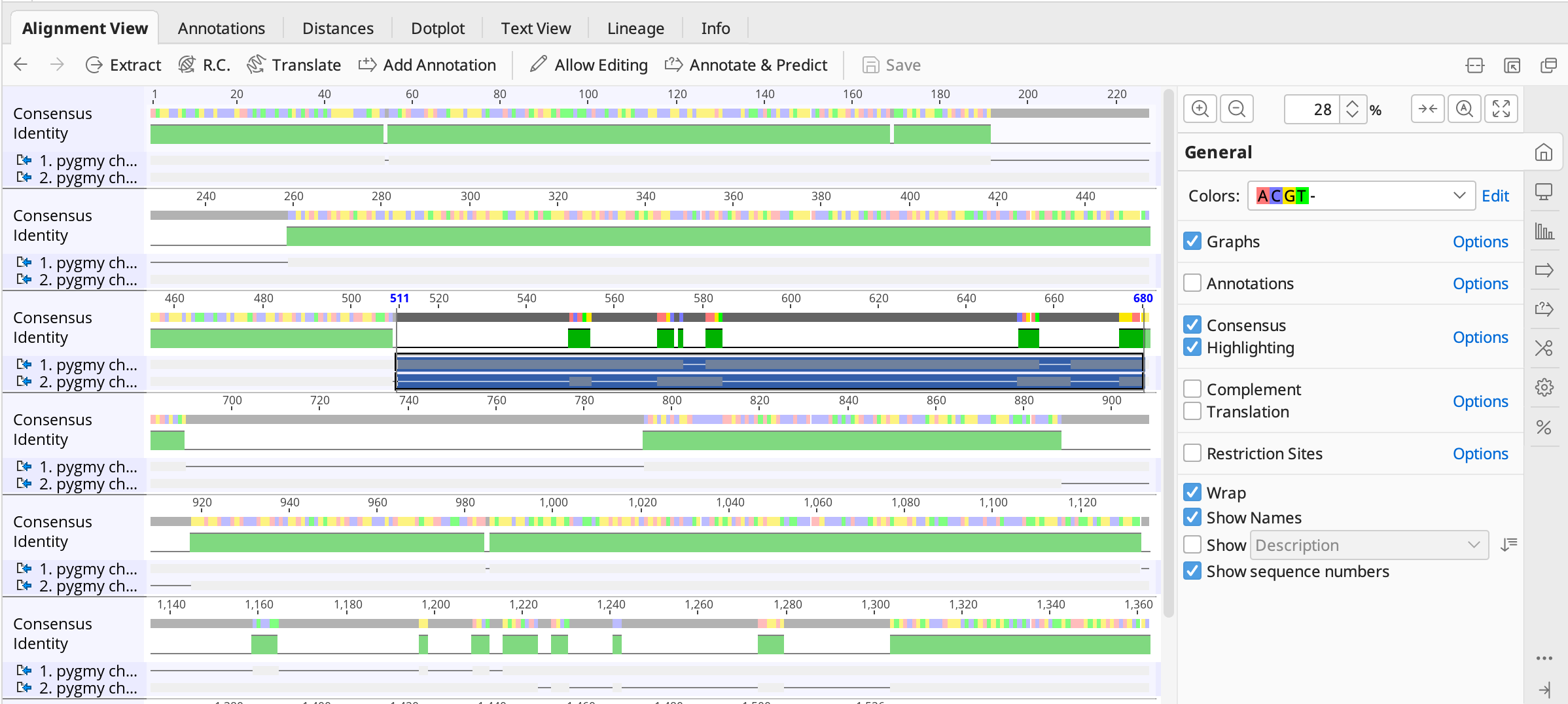
アライメントは数学的に最適ですが、生物学的に必ずしも最適とは限りません。生物学的に意味のあるアライメントであるか注意し、場合によっては設定を変更して最適経路を求める必要があります。




